今回で全5回のR for beginnersの最終回です。
最後は統計解析の醍醐味ですが、「検定と回帰分析」について書きます。
【今日の狙い】
統計学な検定や回帰モデルをRで実行できるようになる。
sample <- read.csv("sample.csv", sep=",", header=T) #attach()を使用して、これからはsampleのみを使用することを宣言する。解除する場合は、detach()関数を使用する。 attach(sample)
1. 相関係数
二つの連続変量同士の関係を探索する
1-1. 基本的な相関係数(ピアソンの相関係数)
#2つの連続量を指定する cor(age, BMI) ## [1] -0.0646569 #複数の連続量に対して、まとめて算出する round(cor(sample[,c(2, 7:15)]), 2) ## age height weight BMI bodyfat sbp dbp bs TC TG ## age 1.00 -0.20 -0.17 -0.06 0.15 -0.05 -0.06 -0.05 0.03 -0.06 ## height -0.20 1.00 0.44 -0.14 -0.39 0.12 0.20 0.17 -0.08 0.16 ## weight -0.17 0.44 1.00 0.83 -0.33 0.14 0.16 0.15 -0.05 0.11 ## BMI -0.06 -0.14 0.83 1.00 -0.12 0.08 0.06 0.06 -0.01 0.02 ## bodyfat 0.15 -0.39 -0.33 -0.12 1.00 -0.09 -0.16 -0.14 0.07 -0.12 ## sbp -0.05 0.12 0.14 0.08 -0.09 1.00 0.03 0.05 -0.01 0.05 ## dbp -0.06 0.20 0.16 0.06 -0.16 0.03 1.00 0.06 -0.03 0.07 ## bs -0.05 0.17 0.15 0.06 -0.14 0.05 0.06 1.00 -0.04 0.05 ## TC 0.03 -0.08 -0.05 -0.01 0.07 -0.01 -0.03 -0.04 1.00 0.00 ## TG -0.06 0.16 0.11 0.02 -0.12 0.05 0.07 0.05 0.00 1.00 #相関のプロットを描く(多くの情報を与えてくれるので便利である。一方で、細かすぎて多変量には不向きである。もう少し少ない変数同士での実行が好ましい。) library(psych) pairs.panels(sample[,c(2, 7:15)])

#相関のカラープロットを描く(いい感じで傾向が掴める) library(qgraph)
#cor.plotでcorによって求める相関係数をヒートマップにする。(numbers=Tで数字も記入する) cor.plot(cor(sample[,c(2,7:15)]), numbers=T)
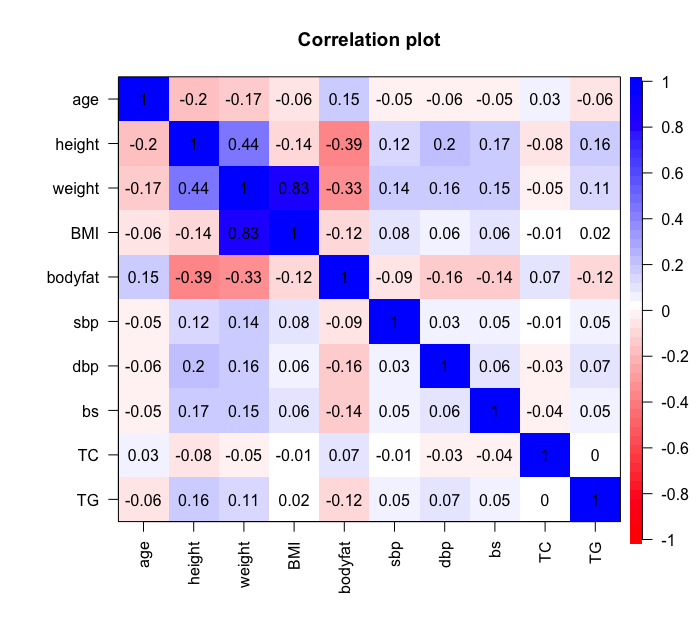
#統計学的にrが0でないか否かの検定 cor.test(age, BMI, method="pearson") ## ## Pearson's product-moment correlation ## ## data: age and BMI ## t = -3.5376, df = 2981, p-value = 0.00041 ## alternative hypothesis: true correlation is not equal to 0 ## 95 percent confidence interval: ## -0.10031247 -0.02883547 ## sample estimates: ## cor ## -0.0646569
1-2. スピアーマンの相関係数(外れ値があっても頑健な方法)
#2つの連続量を指定する cor(age, TG) ## [1] -0.0632198 #統計学的にrが0でないか否かの検定 cor.test(age, TG, method="spearman") ## ## Spearman's rank correlation rho ## ## data: age and TG ## S = 4564100000, p-value = 0.08356 ## alternative hypothesis: true rho is not equal to 0 ## sample estimates: ## rho ## -0.0316874 ## ## 警告メッセージ: ## cor.test.default(age, TG, method = "spearman") で: ## タイのため正確な p 値を計算することができません
1-3. 偏相関係数
ある2変数間(x1とx2)の関係を相関係数で評価しようとすると、しばしばそこには3つ目の変数(x3)が影響を与えている場合がある。その影響を除去して、x1とx2との相関を評価しようとしているのが偏相関係数(partial correlation)である。
ppcorというパッケージをインストールして、その中のpcorで偏相関係数を求めることが出来る。
library(ppcor) #データセット内の連続変数全体での偏相関係数の結果をpcor.resに格納する。 pcor.res <- pcor(sample[,c(2,7:15)]) #pcor.res$estimateで相関係数を返す。 round(pcor.res$estimate, 2) ## age height weight BMI bodyfat sbp dbp bs TC TG ## age 1.00 0.00 -0.03 0.02 0.06 -0.01 0.00 -0.01 0.01 -0.03 ## height 0.00 1.00 0.98 -0.98 -0.01 0.00 0.03 0.00 0.01 0.03 ## weight -0.03 0.98 1.00 0.99 -0.05 0.02 0.00 0.02 -0.02 -0.01 ## BMI 0.02 -0.98 0.99 1.00 0.04 -0.01 0.00 -0.01 0.02 0.01 ## bodyfat 0.06 -0.01 -0.05 0.04 1.00 -0.03 -0.07 -0.06 0.04 -0.06 ## sbp -0.01 0.00 0.02 -0.01 -0.03 1.00 0.00 0.02 0.01 0.02 ## dbp 0.00 0.03 0.00 0.00 -0.07 0.00 1.00 0.02 -0.01 0.03 ## bs -0.01 0.00 0.02 -0.01 -0.06 0.02 0.02 1.00 -0.02 0.02 ## TC 0.01 0.01 -0.02 0.02 0.04 0.01 -0.01 -0.02 1.00 0.02 ## TG -0.03 0.03 -0.01 0.01 -0.06 0.02 0.03 0.02 0.02 1.00 #pcor.res$p.valueでそれぞれのp値を返す。 round(pcor.res$p.value, 2) ## age height weight BMI bodyfat sbp dbp bs TC TG ## age 0.00 0.97 0.12 0.27 0.00 0.45 0.83 0.77 0.51 0.17 ## height 0.97 0.00 0.00 0.00 0.49 0.93 0.17 0.85 0.48 0.15 ## weight 0.12 0.00 0.00 0.00 0.00 0.26 0.87 0.28 0.22 0.72 ## BMI 0.27 0.00 0.00 0.00 0.05 0.52 0.83 0.49 0.24 0.64 ## bodyfat 0.00 0.49 0.00 0.05 0.00 0.14 0.00 0.00 0.02 0.00 ## sbp 0.45 0.93 0.26 0.52 0.14 0.00 0.93 0.20 0.66 0.28 ## dbp 0.83 0.17 0.87 0.83 0.00 0.93 0.00 0.32 0.58 0.10 ## bs 0.77 0.85 0.28 0.49 0.00 0.20 0.32 0.00 0.19 0.29 ## TC 0.51 0.48 0.22 0.24 0.02 0.66 0.58 0.19 0.00 0.27 ## TG 0.17 0.15 0.72 0.64 0.00 0.28 0.10 0.29 0.27 0.00
#偏相関係数の結果をカラープロットする。 cor.plot(pcor.res$estimate, numbers=T)

#その他にも3つの変数を設定して、偏相関係数を求めることもできる。 #この場合は収縮期血圧と血糖値との関係を肥満指標であるBMIで調整したものと考えることが出来る。 pcor.test(sbp, bs, BMI) ## estimate p.value statistic n gp Method ## 1 0.04976019 0.006571088 2.719747 2983 1 pearson
2. t検定(パラメトリックな検定)
二群間のある値(連続量)に差があるか検定する
#等分散を仮定する場合(Studentのt検定を実行したい場合)はvar.equal=Tとして指定する必要がある。 #デフォルトでは、分散が二群で異なる場合に行うWelchのt検定を実行する設定になっている。 t.test(BMI ~ sex) ## ## Welch Two Sample t-test ## ## data: BMI by sex ## t = -11.938, df = 2866.4, p-value < 2.2e-16 ## alternative hypothesis: true difference in means is not equal to 0 ## 95 percent confidence interval: ## -2.127647 -1.527324 ## sample estimates: ## mean in group Female mean in group Male ## 22.97017 24.79765 boxplot(BMI ~ sex, xlab="Sex", ylab="kg/m2")

3. Wilcoxonの順位和検定(Mann-WhitneyのU検定ともいう。ノンパラメトリックな検定)
#pairedの設定はFならば順位和検定、Tならば符号順位和(対応のある2群のノンパラメトリック検定)となる。 wilcox.test(TG ~ sex, paired=F) ## ## Wilcoxon rank sum test with continuity correction ## ## data: TG by sex ## W = 868290, p-value < 2.2e-16 ## alternative hypothesis: true location shift is not equal to 0 ## boxplot(TG ~ sex, xlab="Sex", ylab="mg/dl")

4. 一元配置分散分析(Analysis of variance: ANOVA)
三群以上のある値(連続量)に差があるか検定する
4.1 aov関数を用いた場合
result <- aov(BMI ~ exercise) summary(result) ## Df Sum Sq Mean Sq F value Pr(>F) ## exercise 2 371 185.43 9.816 5.64e-05 *** ## Residuals 2980 56295 18.89 ## --- ## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
4.2 anova()関数と線形回帰モデルを実行するlm()関数を用いた場合
lm <- lm(BMI ~ exercise) anova(lm) ## Analysis of Variance Table ## ## Response: BMI ## Df Sum Sq Mean Sq F value Pr(>F) ## exercise 2 371 185.432 9.8159 5.636e-05 *** ## Residuals 2980 56295 18.891 ## --- ## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
5. クラスカルウォリス検定(kruskal-Walis test)
対応のある三群以上のある値(連続量)に差があるか検定する。(対応のある3群以上のノンパラ検定はFiredman testを実行する。)
kruskal.test(TG ~ exercise, data=sample) ## ## Kruskal-Wallis rank sum test ## ## data: TG by exercise ## Kruskal-Wallis chi-squared = 4.4202, df = 2, p-value = 0.1097
6. 多重比較(multiple comparison)
ANOVAやクラスカルウォリス検定で有意になった場合に、さらにどこの群間に差が見られるのかを検討する手法(今回はTukey-KrammerとSteel-Dwass検定を例として表示する。その他の情報についてはwebの情報を参照されたい。)
#Tukey-Krammer(テューキー・クレイマー)検定を実行する。(一般的にはテューキー法やTukey Honestly significant Different (Tukey's HSD)testなどと呼ばれる) TukeyHSD(aov(BMI ~ exercise)) ## Tukey multiple comparisons of means ## 95% family-wise confidence level ## ## Fit: aov(formula = BMI ~ exercise) ## ## $exercise ## diff lwr upr p adj ## low-high 0.06284613 -0.5928998 0.7185921 0.9725429 ## middle-high 0.75697565 0.1055530 1.4083983 0.0177742 ## middle-low 0.69412951 0.3005685 1.0876906 0.0001077 plot(TukeyHSD(aov(BMI ~ exercise))) #Steel-Dwassについてはパッケージなどでは公開されておらず、群馬大学青木先生によって公開されているSteel.Dwassという自作の関数を使用する #これは漸近的なものであり、NSM3パッケージの中にもsteel-Dwass検定を行うものがあることをここで記載したい。 Steel.Dwass <- function(data,group) { OK <- complete.cases(data, group) data <- data[OK] group <- group[OK] n.i <- table(group) ng <- length(n.i) t <- combn(ng, 2, function(ij) { i <- ij[1] j <- ij[2] r <- rank(c(data[group == i], data[group == j])) R <- sum(r[1:n.i[i]]) N <- n.i[i]+n.i[j] E <- n.i[i]*(N+1)/2 V <- n.i[i]*n.i[j]/(N*(N-1))*(sum(r^2)-N*(N+1)^2/4) return(abs(R-E)/sqrt(V)) }) p <- ptukey(t*sqrt(2), ng, Inf, lower.tail=FALSE) result <- cbind(t, p) rownames(result) <- combn(ng, 2, paste, collapse=":") return(result) } #Steel.DwassはSteel.Dwass(目的の値, 数値に変換したグループ変数)で実行できる Steel.Dwass(TG, as.numeric(exercise)) ## t p ## 1:2 1.631953 0.2321546 ## 1:3 0.502737 0.8699921 ## 2:3 1.781280 0.1758568

Steel.dwassに関してはこちらの記事を御覧ください。
jojoshin.hatenablog.com
7. カイ二乗検定(Chi-squared test)
カイ二乗検定は、カテゴリー変数の解析においては非常にpopularな解析方法である。一変量の場合にその観察分布が期待される分布に従っているかを検討する場合には、「適合度の検定」と呼ばれ、また2つのカテゴリ変数について、その関連性を検討する場合には「独立性の検定」と呼ばれている。今回はより使用頻度の高いと考えられる独立性の検定について例を示して、実行する(イエーツの連続性の補正も含む)。
#今回は性別とBMI25以上か否かの2つのカテゴリー変数間に関連があるかをカイ二乗検定で確かめる。 #まず、二つの変数を用いて、それぞれの特性を持つ人数を示す分割表を作成する。 table1 <- table(sex, BMI01) plot(sex ~ BMI01) #chisq.test()で実行できる。 chisq.test(table1) ## ## Pearson's Chi-squared test with Yates' continuity correction ## ## data: table1 ## X-squared = 60.115, df = 1, p-value = 8.946e-15

8. Fisherの正確検定(Fisher's exact test)
カイ二乗検定に関連して、カテゴリ変数の解析法の一つであるFisherの正確性検定も紹介する。特に2つのカテゴリ変数の関連性を検討するという目的は共通であるが、セル内の生起数が少ない場合にはFisherの正確検定を推奨されている。
#カイ二乗検定と同様に分割表を作成する。今回は運動習慣とBMI25以上か否かの2つのカテゴリー変数間に関連があるかを検討する。 table2 <- table(exercise, BMI01) fisher.test(table2) ## ## Fisher's Exact Test for Count Data ## ## data: table2 ## p-value = 0.007866 ## alternative hypothesis: two.sided
ここからは回帰モデルを実行する。
回帰分析は、「ある値がa変化する時、目的の値がどれだけ変化するか」を検討するモデル・手法である。
9. 線形回帰モデル(linear regression model)
9-1. 線形回帰モデル(linear regression model)単変量
まずは結果をresultに格納する
result <- lm(HDLC ~ age)
names関数でresult内の内容を見る
names(result) ## [1] "coefficients" "residuals" "effects" "rank" "fitted.values" ## [6] "assign" "qr" "df.residual" "xlevels" "call" ## [11] "terms" "model"
resultを要約する
summary関数はモデルの情報を表示し、残差、係数、標準誤差と各変数のp値、自由度、モデル自体のp値とF統計値を含んでいる。
summary(result) ## ## Call: ## lm(formula = HDLC ~ age) ## ## Residuals: ## Min 1Q Median 3Q Max ## -44.558 -9.636 -0.495 9.083 48.817 ## ## Coefficients: ## Estimate Std. Error t value Pr(>|t|) ## (Intercept) 51.53025 1.35012 38.167 < 2e-16 *** ## age 0.15617 0.02654 5.884 4.45e-09 *** ## --- ## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 ## ## Residual standard error: 13.84 on 2981 degrees of freedom ## Multiple R-squared: 0.01148, Adjusted R-squared: 0.01115 ## F-statistic: 34.62 on 1 and 2981 DF, p-value: 4.455e-09 #Call: モデルの式 #Residuals: 残差の四分位 #Coefficients: 傾きと切片(係数が0であるか否かのt検定とそのp値) #Residual standard error: 残差の標準誤差 #Multiple R-squared: 決定係数 #Adjusted R-squared: 自由度調整済み決定係数 #F-statistic: F比 #p-value: その検定結果
係数のみを取り出し、プロットする
result$coefficients ## (Intercept) age ## 51.530255 0.156171
その他のresultに関連するコマンド(結果省略)
fitted(result) resid(result) predict(result)
9-2. 線形回帰分析(linear regression model)多変量
シンプルな例
#HDLCを性別と年齢で説明するモデル result <- lm(HDLC ~ age + sex)
#結果の解釈は年齢についていえば、年齢が1つ増えるのに対して、HDLは0.01増えることを示している(有意な関連ではない)。性別についてはFemaleに対してMaleであれば、10.1減少することを示し、性別とHDLとの関係は年齢の影響を固定しても(調整しても)有意であることがわかる。 summary(result) ## ## Call: ## lm(formula = HDLC ~ age + sex) ## ## Residuals: ## Min 1Q Median 3Q Max ## -40.431 -8.573 0.441 8.654 44.639 ## ## Coefficients: ## Estimate Std. Error t value Pr(>|t|) ## (Intercept) 62.66711 1.38472 45.256 <2e-16 *** ## age 0.01415 0.02592 0.546 0.585 ## sexMale -10.10510 0.50513 -20.005 <2e-16 *** ## --- ## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 ## ## Residual standard error: 13 on 2980 degrees of freedom ## Multiple R-squared: 0.1285, Adjusted R-squared: 0.1279 ## F-statistic: 219.7 on 2 and 2980 DF, p-value: < 2.2e-16
説明変数が2つ以上ある場合
result <- lm(HDLC ~ age + sex + BMI + alcohol + exercise + smoking) #性別とexerciseの少ない群で関連があった。性別は女性に比べて、男性で有意にHDL値が低い。また、運動量の少ない人も最も多い群に比べて、-1.6ほど低値になることを示唆している。 summary(result) ## ## Call: ## lm(formula = HDLC ~ age + sex + BMI + alcohol + exercise + smoking) ## ## Residuals: ## Min 1Q Median 3Q Max ## -40.551 -8.803 0.068 8.603 45.541 ## ## Coefficients: ## Estimate Std. Error t value Pr(>|t|) ## (Intercept) 63.41764 2.24034 28.307 <2e-16 *** ## age 0.01391 0.02591 0.537 0.5913 ## sexMale -10.37739 0.57522 -18.041 <2e-16 *** ## BMI 0.02438 0.05585 0.436 0.6626 ## alcohollow 0.12193 0.66143 0.184 0.8538 ## alcoholmiddle 0.49092 0.66344 0.740 0.4594 ## exerciselow -1.68100 0.83726 -2.008 0.0448 * ## exercisemiddle -0.70250 0.83561 -0.841 0.4006 ## smokingever 0.20592 0.94712 0.217 0.8279 ## smokingnever -0.71997 0.91151 -0.790 0.4297 ## --- ## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 ## ## Residual standard error: 12.99 on 2973 degrees of freedom ## Multiple R-squared: 0.1314, Adjusted R-squared: 0.1288 ## F-statistic: 49.99 on 9 and 2973 DF, p-value: < 2.2e-16 #係数の関係性(影響の向きと大きさ)を示すのが、coefplotパッケージのcoefplot()関数である。それを使ってこの結果を描画する。 library(coefplot) coefplot(result)

10. 一般化線形回帰分析(generalized linear regression model)
目的変数は正規分布しているものばかりではない。よく医学の分野で目にするのは疾患の発症の有無や基準値以上もしくはそれ以下などの01データが代表例である。今回はそのような01データを目的変数に取る解析とある事象の発生確率が極めて低い分布いわゆるポアソン分布を目的変数とする解析の例を紹介する。
10-1. ロジスティック回帰分析(logistic regression model)
BMIの25以上or25未満を目的変数として、年齢、アルコールの摂取頻度(3カテゴリ)、性別の3つの説明変数を持つモデルでロジスティック回帰分析をする。
#glm()が一般化線形回帰分析の意味で、その結果をresultに格納する。 #本日の7.カイ二乗検定では性別とBMI01との直接的な関連を探索したが、ここでは性別とBMI01の関係を年齢とアルコール摂取量の変数で補正しているモデルとも捉えることができる。(しばしば、手始めにカイ二乗検定等であたりをつけて、多変量で交絡要因を補正したモデルで検討することがある。) result <- glm(BMI01 ~ age + alcohol + sex, data=sample) #結果を見る。性別が有意な関連を示すことがわかる。 summary(result) ## ## Call: ## glm(formula = BMI01 ~ age + alcohol + sex, data = sample) ## ## Deviance Residuals: ## Min 1Q Median 3Q Max ## -0.4941 -0.3389 -0.3007 0.5554 0.7249 ## ## Coefficients: ## Estimate Std. Error t value Pr(>|t|) ## (Intercept) 0.3748633 0.0543556 6.896 6.48e-12 *** ## age -0.0014894 0.0009514 -1.565 0.118 ## alcohollow 0.0062872 0.0242589 0.259 0.796 ## alcoholmiddle 0.0295496 0.0243378 1.214 0.225 ## sexMale 0.1343819 0.0199625 6.732 2.00e-11 *** ## --- ## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 ## ## (Dispersion parameter for gaussian family taken to be 0.2275686) ## ## Null deviance: 692.78 on 2982 degrees of freedom ## Residual deviance: 677.70 on 2978 degrees of freedom ## AIC: 4056.6 ## ## Number of Fisher Scoring iterations: 2 #ロジスティック回帰分析の一つの有効な指標としてオッズ比がある。 #それらはe^回帰係数で簡単に求めることができる。 exp(result$coefficients) ## (Intercept) age alcohollow alcoholmiddle sexMale ## 1.4547925 0.9985117 1.0063070 1.0299906 1.1438296 #さらにオッズ比の95%信頼区間とp値を加えた出力法を紹介する。 ModelName <- result alpha <- 0.05 x <- summary(ModelName) y <- confint(ModelName, level=1-alpha) OR <- round(exp(x$coefficients[,1]), 2) LowerCL <- round(exp(y[,1]), 2) UpperCL <- round(exp(y[,2]), 2) pval <- round(x$coefficients[,4], 3) z <- cbind(OR, LowerCL, UpperCL, pval) #年齢とアルコール摂取量の変数を補正しても、性別が有意な関連を示し、Femaleと比較してMaleでBMI25以上のリスク上昇があると考えられる。 z ## OR LowerCL UpperCL pval ## (Intercept) 1.45 1.31 1.62 0.000 ## age 1.00 1.00 1.00 0.118 ## alcohollow 1.01 0.96 1.06 0.796 ## alcoholmiddle 1.03 0.98 1.08 0.225 ## sexMale 1.14 1.10 1.19 0.000 #sampleはここまでで使用を終わるので、detachする detach(sample)
10-2. ポアソン回帰分析(poisson regression model)
ポアソン回帰は一定時間内に発生した事象を数えたデータ(いわゆるカウントデータ)の代表的な解析であり、発生事象とその要因について探索する回帰モデルである。カウントデータ解析での目的変数はもちろん発生数であり、その分布はしばしば下記のように1)離散であり、2)非負かつ3)標本分散と平均がほとんど等しいポアソン分布をとることからこのような回帰分析が行われる。医学データでは交通事故の発生数、一定期間の症例の発症数などポアソン回帰分析を使用する事例もいくつもある。
#サンプルデータ(ある地域における2017年1月のMIによる死亡数と気象要因との関連)を作成する。 MI <- c(0, 1, 1, 2, 3, 1, 1, 5, 1, 4, 0, 0, 1, 1, 1, 3, 5, 2, 1, 0, 1, 2, 0, 1, 1, 2, 1, 4, 2, 3, 4) Temp <- c(9.0, 6.2, 8.6, 5.5, 2.4, 10.0, 7.2, 1.2, 4.6, 2.9, 6.7, 9.2, 6.2, 7.2, 8.1, 4.3, 2.3, 6.7, 7.7, 9.4, 8.2, 4.4, 7.8, 6.9, 5.7, 4.2, 6.4, 3.2, 5.5, 6.2, 3.2) Wind <- c(3, 4, 5, 3, 2, 6, 4, 3, 2, 4, 3, 5, 5, 6, 2, 9, 1, 3, 2, 5, 4, 2, 2, 2, 6, 2, 1, 5, 3, 3, 2) Humid <- c(60, 62, 66, 98, 54, 33, 45, 55, 78, 82, 64, 74, 51, 66, 88, 43, 42, 39, 60, 59, 62, 64, 77, 71, 78, 42, 91, 52, 53, 68, 55) Month <- rep(1, 31) Day <- 1:31 MIsample <- data.frame(MI, Temp, Wind, Humid, Month, Day) head(MIsample) ## MI Temp Wind Humid Month Day ## 1 0 9.0 3 60 1 1 ## 2 1 6.2 4 62 1 2 ## 3 1 8.6 5 66 1 3 ## 4 2 5.5 3 98 1 4 ## 5 3 2.4 2 54 1 5 ## 6 1 10.0 6 33 1 6 #心筋梗塞による死亡数の分布(ポアソン分布の例) hist(MI) #心筋梗塞による死亡数と気象変数(気温、湿度、風速)との関連をポアソン回帰(familly=poissonと指定する)で検討する result <- glm(MI ~ Temp + Wind + Humid, data=MIsample, family=poisson) summary(result) ## ## Call: ## glm(formula = MI ~ Temp + Wind + Humid, family = poisson, data = MIsample) ## ## Deviance Residuals: ## Min 1Q Median 3Q Max ## -1.47267 -0.41522 0.01451 0.36422 1.34112 ## ## Coefficients: ## Estimate Std. Error z value Pr(>|z|) ## (Intercept) 2.359459 0.637094 3.703 0.000213 *** ## Temp -0.301399 0.065025 -4.635 3.57e-06 *** ## Wind 0.028758 0.073796 0.390 0.696766 ## Humid -0.005396 0.009748 -0.554 0.579897 ## --- ## Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 ## ## (Dispersion parameter for poisson family taken to be 1) ## ## Null deviance: 39.158 on 30 degrees of freedom ## Residual deviance: 12.244 on 27 degrees of freedom ## AIC: 85.046 ## ## Number of Fisher Scoring iterations: 5 #結果の解釈としてはやはり気温が密接に関与していることが見て取れる。 #具体的には、気温が1℃上昇すると、MIの死亡数は-0.3件となる。

11. モデルの選択(Model selection)
できるだけ最大化対数尤度が大きいあてはまりのよいモデル、できるだけパラメーター数が少ない簡単なモデルを選ぶことがモデルの構築では求められる。今回はその過程を少し説明する。
パッケージMASSのstepAIC()関数でモデリングの指標であるAIC(赤池情報量)に従って、変数選択をする。
library(MASS) #再びsampleデータに戻って、自らによってBMI01を説明するモデル(フルモデル)を構築する。 full.model <- glm(BMI01 ~ age + sex + alcohol + exercise + smoking + sbp + bs + TC + TG, data = sample) #AICが減っている間は1つずつ変数を減らしていく。そして変数を除去して、AICが大きくなる場合はその前に戻り、そこを最終的なモデル(AICによる最良のモデル)とする。 stepAIC(full.model) #結果は省略する
補足. 多変量モデルの記述例
y ~ x #yはxによって説明される y ~ x1 + x2 + ... + xn #yはx1 + x2 + ... + xnを説明変数とする y ~ . #y以外の変数全てを説明変数とする y ~ . -x #xは予測項には含めない y ~ x1 + x2 + ... + xn -1 #定数項は予測項には含めない y ~ x1*x2 + ... + xn #x1とx2の交互作用項も含める
##参考資料
###[1] 馬場真哉(2015)『平均・分散から始める一般化線形モデル入門』プレアデス出版
###[2] 久保拓弥(2012)『データ解析のための統計モデリング入門――一般化線形モデル・階層ベイズモデル・MCMC (確率と情報の科学)』岩波出版
最後に
これが5回シリーズのR初心者のための資料になります。初めてこのような資料を作成しておりますので、何か間違い等あれば是非コメントを通じてご連絡お願い致します。
20160613
RF